Difference between revisions of "ProjectYeast"
Sbprm2015 1 (talk | contribs) |
Sbprm2015 1 (talk | contribs) |
||
| (52 intermediate revisions by the same user not shown) | |||
| Line 1: | Line 1: | ||
| − | + | ||
| + | |||
| + | '''MODELING GRADIENT FORMATION IN THE FISSION YEAST''' | ||
| + | |||
---- | ---- | ||
| − | Escher Cécile, Steffe Perrine, Spataro Sofia, Murseli Besa | + | '''Escher Cécile, Steffe Perrine, Spataro Sofia, Murseli Besa''' |
| − | Superviseur: Hersch Micha | + | '''Superviseur: Hersch Micha''' |
---- | ---- | ||
| − | |||
| − | 1 | + | '''1 - Introduction''' |
| − | + | ||
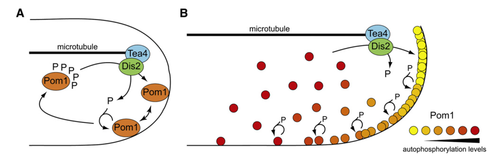
| − | [[File: | + | Chez la levure ''Schizosaccharomyces pombe'', le gradient de concentration de la protéine Pom1 est déterminant dans le positionnement du plan de la division cellulaire. Pom1 régule négativement un activateur de l'entrée en mitose: la protéine kinase Cdr2, qui se situe au niveau de l'équateur de la cellule. Son transport aux pôles de la cellule par la protéine Tea4, qui se trouve sur les microtubules du cytosquelette de la cellule, crée la formation du gradient de Pom1. Une fois au pôle, Pom1 se liera au lipide de la membrane via des interactions électrostatiques et par son activité de kinase, Pom1 diffusera le long de la membrane en alternat phosphorylation et déphosphorylation sur ses différent sites cibles. |
| + | Lors de ce projet, le phénomène de la diffusion de Pom1 a été abordé en étudiant, sur la base de réflexions mathématiques, la variation du domaine de la protéine de Cdr2 en fonction de celle de Tea4 et Pom1. Le clustering de Pom1 (rassemblement de Pom1 en agrégat) a été dans un second temps au centre de notre étude. | ||
| + | [[File:Gradient_.png|500px|thumb|center|Formation du gradient de Pom1]] | ||
| + | |||
| + | '''2 - Phosphorylation de Pom1: modèle ''cis'' ou ''trans''?''' | ||
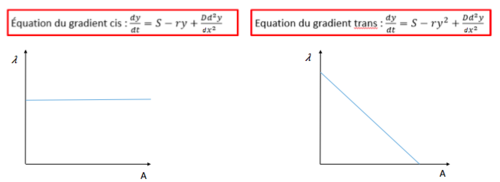
| + | Dans un premier temps, nous nous sommes intéressé au mode de phosphorylation de Pom1. Une protéine pom1 phosphoryle-t-elle ses propres sites ou plutôt ceux de ses voisines ? Dans le premier cas on parlerait de modèle en ''trans'' et dans le second, de modèle en ''cis''. Pour répondre à cette question, nous avons analysé avec R les profiles de l'intensité de Pom1 le long de la membrane pour chercher de comprendre si λ; la distance à laquelle la concentration de Pom1 diminue d'un facteur donné, dépend de la valeur A; la quantité initiale de Pom1 au temps 0 au pôle. S'il n'y a pas de corrélation entre λ et A le modèle de phosphorylation est dit en ''cis'' et au contraire, si corrélation il y a, le modèle est dit en ''trans'' et on est en présence d’auto-phosphorylation. | ||
| + | Pour obtenir ces modèles nous avons travaillé avec des équations différentielles: | ||
| + | [[File:cistrans2.png|500px|thumb|center|Modèles ''cis'' et ''trans'']] | ||
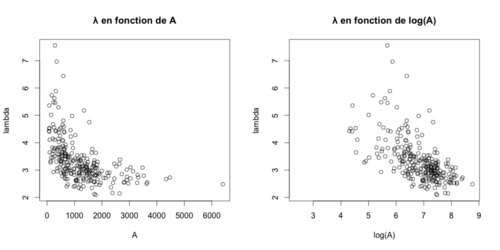
| + | Voici les résultats: | ||
| + | [[File:Resultspom1.png|500px|thumb|center|λ en fonction de A et de log(A)]] | ||
| + | Nous avons pu conclure que le mode de phosphorylation de Pom1 était en ''trans'' et donc, que chaque protéine de Pom1 phosphoryle ses propres sites. | ||
| + | '''3 - Localisation de Cdr2 en fonction de Tea4''' | ||
| + | Cette partie porte sur l'étude de la localisation de Cdr2 en fonction de la variabilité de Tea4 aux extrémités de la cellule. | ||
| + | Les données que nous avions pour cela ne nous permettait pas d'étudier directement la variation du domaine Cdr2 en fonction de celle de la quantité de Tea4. Cependant, nous en avions pour Tea4 et Pom1 qui provenaient des mêmes cellules et pour Pom1 avec Cdr2 pour d'autres mêmes cellules. En prouvant qu'il y a une corrélation entre la variabilité de la quantité de Pom1 au pôle avec celle de Tea4 et qu'il y également une corrélation entre la variabilité du domaine de Cdr2 au centre de la cellule avec la variabilité de concentration de Pom1 au pôle, nous conclure que l'étendu du domaine de Cdr2 est corrélé avec la concentration de Tea4 aux pôles. | ||
| + | '''Tea 4 et Pom1''' | ||
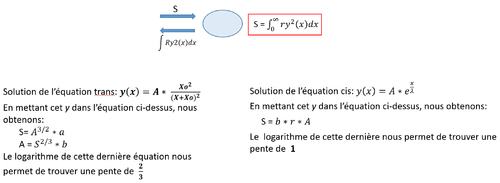
| + | Nous avons, dans R, représenté graphiquement les valeurs de l'amplitude A de Pom1 (comme dit plus haut, A est la quantité initiale de Pom1 au temps 0 aux pôles) en fonction de l'amplitude S de tea4 (S est la quantité initiale de Tea4 au temps 0 aux pôles). S'il n'y a pas de corrélation entre les deux, nous nous attendons à une pente de 0 et au contraire, s'il y a corrélation, nous aurons dans le cas ''trans'' une pente de 2/3 et dans le cas ''cis'' une pente de 1. Ces pentes ont été trouvé à l'aide des équations suivantes: | ||
| + | [[File:Tea4Pom1.png|500px|thumb|center|Corrélation: Pente en cas ''trans'' et cas ''cis'']] | ||
| + | Résultats: | ||
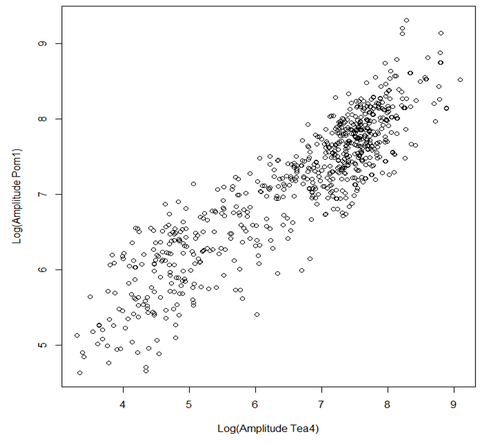
| + | [[File:GrapheTea4Pom1.png|500px|thumb|center|A en fonction de S]] | ||
| + | La pente de cette droite est 0.63 soit environ 2/3. Sur la base de ce résultat, nous avons conclu qu'il y avait une corrélation entre la variabilité de quantité de Pom1 aux pôles avec celle de Tea4 et avons confirmé que nous avions bien en modèle en ''trans''. | ||
| + | '''Pom1 et Cdr2''' | ||
| + | Nous avons, ici aussi, représenté graphiquement dans R les différentes longueur du domaine de Cdr2 en fonction de l'amplitude moyenne de A. | ||
| + | Voici ce que nous avons obtenu: | ||
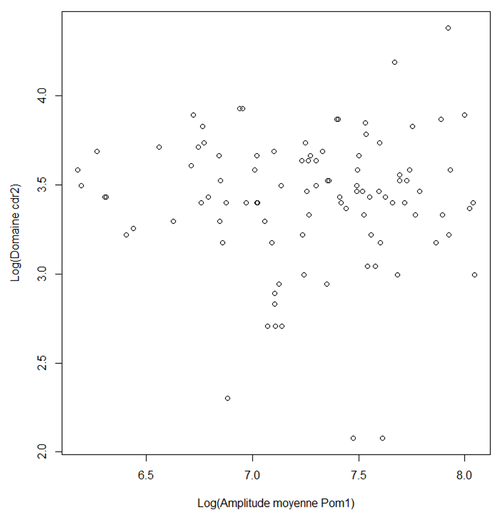
| + | [[File:GrapheCdr2Pom1.png|500px|thumb|center|Taille du domaine de Cdr2 en fonction de la concentration moyenne de Pom1 aux pôles]] | ||
| + | Pour trouver la pente de cette droite nous avons utiliser la régression linéaire. Nous avons une pente de 0 avec une probabilité associée au test (P-value) de 0.95. Ceci ne nous permet pas de rejeter significativement l'hypothèse que la taille du domaine de cdr2 est indépendante de la concentration de pom1 se trouvant aux pôles. Nous ne pouvons donc pas dire que dommaine de Cdr2 est corrélé avec la quantité de Pom1.Ce résultat n'est pas du tout celui auquel nous nous attendions car il est contraire à ce que la théorie dit. | ||
| + | L'effet que Pom1 a sur la taille du domaine de Pom1 est un effet petit ce qui pourrait expliquer pourquoi il est difficile à expliquer. De plus, l'effet des variations d'amplitude de Pom1 au niveau du pôle de la cellules sont atténuées au niveau du centre de la cellule. Cela permet à la cellule d'être précise pour déterminer le milieu de la cellule, même si la quantité de Pom1 aux extrémités de le cellule est très variable de cellule à cellule. C'est ce qu'on appelle le "Buffering" qui peut se traduire par "atténuation". | ||
| + | '''4 - Clustering de Pom1''' | ||
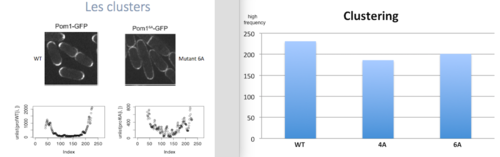
| + | Dans cette partie on va étudier la relation entre la formation de clusters (agrégats de Pom1) et la phosphorylation de Pom1. On a la levure wild type et troi types de mutants: 2A, avec deux sites de phosphorylation mutés, 4A, qui en a quatre, et 6A, qui en a six. Comme première chose, on a voulu quantifier les pics des profiles des mutants, pour comprendre si la phosphorylation a un rôle dans le clustering. On a donc travaillé avec la transformée de Fourier, que c’est une somme infinie des fonctions sinus et cosinus ayant comme argument la fréquence. Cette analyse dans R correspond à la fonction ''spectrum''. Au final on a comparés les vecteur avec les différentes hautes fréquences à l’aide d’un test de Student. Les résultats qu’on a obtenus sont des p valeurs significatives, avec un seuil de 0.05, sauf pour le mutant 2A, qui ce n’est pas significativement différent du WT. On a donc conclu que les mutants 4A et 6A sont significativement différents du WT et font moins de clusters. La phosphorylation joue donc un rôle dans le clustering, car moins il peut se phosphoryler, moins il fait des clusters. | ||
| + | [[File:clust.png|500px|thumb|center|Gauche: photos et profiles du WT et du mutant 6A. Droite: Formation de clusters par phénotype]] | ||
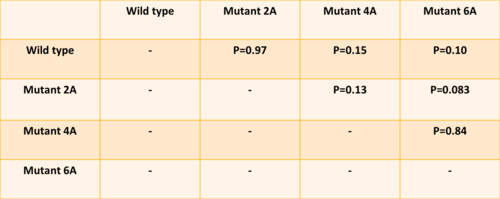
| + | Dans un deuxième temps, on a cherché à comprendre quelle implication a le clustering dans la formation du gradient. Pour faire ça on a calculé la quantité de Pom1 dans la cellule. Dans R on s’est servi de la fonction cortical.pom1, qui nous a donné notre superviseur, qui tient en compte la courbe de la cellule. Enfin, on a appliqué aussi des t-tests. On a obtenu des p valeurs pas significatives, donc il n'y a pas plus de Pom1 à la membrane dans les mutants par rapport au WT. | ||
| + | [[File:ttest.png|500px|thumb|center|P valeurs des t-tests: comparaison de la quantité de Pom1 à la membrane]] | ||
| + | On a expliqué ça par le fait que peut-être que les sites de phosphorylation sont utiles pour s’attacher et se détacher de la membrane. | ||
| + | '''5 - Conclusions''' | ||
| − | + | Notre première conclusion a été que le mode de phosphorylation de Pom1 est ''trans''. Deuxièmement, on a trouvé une corrélation entre la quantité de Tea4 et Pom1, mais pas entre Pom1 et Cdr2. Enfin, la formation de clusters dépend de la phosphorylation. Comme certains résultats n’ont pas été significatifs, d’autres expériences sont nécessaires pour répondre à la question. Pendant ce projet on a pu voir comme des modèles mathématiques peuvent permettre de répondre à des questions biologiques. | |
| − | + | Nous remercions chaleureusement notre superviseur [[User:Micha|Micha Hersch]] pour son aide tout au long de ce projet. | |
Latest revision as of 12:52, 28 May 2015
MODELING GRADIENT FORMATION IN THE FISSION YEAST
Escher Cécile, Steffe Perrine, Spataro Sofia, Murseli Besa
Superviseur: Hersch Micha
1 - Introduction
Chez la levure Schizosaccharomyces pombe, le gradient de concentration de la protéine Pom1 est déterminant dans le positionnement du plan de la division cellulaire. Pom1 régule négativement un activateur de l'entrée en mitose: la protéine kinase Cdr2, qui se situe au niveau de l'équateur de la cellule. Son transport aux pôles de la cellule par la protéine Tea4, qui se trouve sur les microtubules du cytosquelette de la cellule, crée la formation du gradient de Pom1. Une fois au pôle, Pom1 se liera au lipide de la membrane via des interactions électrostatiques et par son activité de kinase, Pom1 diffusera le long de la membrane en alternat phosphorylation et déphosphorylation sur ses différent sites cibles. Lors de ce projet, le phénomène de la diffusion de Pom1 a été abordé en étudiant, sur la base de réflexions mathématiques, la variation du domaine de la protéine de Cdr2 en fonction de celle de Tea4 et Pom1. Le clustering de Pom1 (rassemblement de Pom1 en agrégat) a été dans un second temps au centre de notre étude.
2 - Phosphorylation de Pom1: modèle cis ou trans?
Dans un premier temps, nous nous sommes intéressé au mode de phosphorylation de Pom1. Une protéine pom1 phosphoryle-t-elle ses propres sites ou plutôt ceux de ses voisines ? Dans le premier cas on parlerait de modèle en trans et dans le second, de modèle en cis. Pour répondre à cette question, nous avons analysé avec R les profiles de l'intensité de Pom1 le long de la membrane pour chercher de comprendre si λ; la distance à laquelle la concentration de Pom1 diminue d'un facteur donné, dépend de la valeur A; la quantité initiale de Pom1 au temps 0 au pôle. S'il n'y a pas de corrélation entre λ et A le modèle de phosphorylation est dit en cis et au contraire, si corrélation il y a, le modèle est dit en trans et on est en présence d’auto-phosphorylation. Pour obtenir ces modèles nous avons travaillé avec des équations différentielles:
Voici les résultats:
Nous avons pu conclure que le mode de phosphorylation de Pom1 était en trans et donc, que chaque protéine de Pom1 phosphoryle ses propres sites.
3 - Localisation de Cdr2 en fonction de Tea4
Cette partie porte sur l'étude de la localisation de Cdr2 en fonction de la variabilité de Tea4 aux extrémités de la cellule. Les données que nous avions pour cela ne nous permettait pas d'étudier directement la variation du domaine Cdr2 en fonction de celle de la quantité de Tea4. Cependant, nous en avions pour Tea4 et Pom1 qui provenaient des mêmes cellules et pour Pom1 avec Cdr2 pour d'autres mêmes cellules. En prouvant qu'il y a une corrélation entre la variabilité de la quantité de Pom1 au pôle avec celle de Tea4 et qu'il y également une corrélation entre la variabilité du domaine de Cdr2 au centre de la cellule avec la variabilité de concentration de Pom1 au pôle, nous conclure que l'étendu du domaine de Cdr2 est corrélé avec la concentration de Tea4 aux pôles.
Tea 4 et Pom1
Nous avons, dans R, représenté graphiquement les valeurs de l'amplitude A de Pom1 (comme dit plus haut, A est la quantité initiale de Pom1 au temps 0 aux pôles) en fonction de l'amplitude S de tea4 (S est la quantité initiale de Tea4 au temps 0 aux pôles). S'il n'y a pas de corrélation entre les deux, nous nous attendons à une pente de 0 et au contraire, s'il y a corrélation, nous aurons dans le cas trans une pente de 2/3 et dans le cas cis une pente de 1. Ces pentes ont été trouvé à l'aide des équations suivantes:
Résultats:
La pente de cette droite est 0.63 soit environ 2/3. Sur la base de ce résultat, nous avons conclu qu'il y avait une corrélation entre la variabilité de quantité de Pom1 aux pôles avec celle de Tea4 et avons confirmé que nous avions bien en modèle en trans.
Pom1 et Cdr2
Nous avons, ici aussi, représenté graphiquement dans R les différentes longueur du domaine de Cdr2 en fonction de l'amplitude moyenne de A. Voici ce que nous avons obtenu:
Pour trouver la pente de cette droite nous avons utiliser la régression linéaire. Nous avons une pente de 0 avec une probabilité associée au test (P-value) de 0.95. Ceci ne nous permet pas de rejeter significativement l'hypothèse que la taille du domaine de cdr2 est indépendante de la concentration de pom1 se trouvant aux pôles. Nous ne pouvons donc pas dire que dommaine de Cdr2 est corrélé avec la quantité de Pom1.Ce résultat n'est pas du tout celui auquel nous nous attendions car il est contraire à ce que la théorie dit. L'effet que Pom1 a sur la taille du domaine de Pom1 est un effet petit ce qui pourrait expliquer pourquoi il est difficile à expliquer. De plus, l'effet des variations d'amplitude de Pom1 au niveau du pôle de la cellules sont atténuées au niveau du centre de la cellule. Cela permet à la cellule d'être précise pour déterminer le milieu de la cellule, même si la quantité de Pom1 aux extrémités de le cellule est très variable de cellule à cellule. C'est ce qu'on appelle le "Buffering" qui peut se traduire par "atténuation".
4 - Clustering de Pom1
Dans cette partie on va étudier la relation entre la formation de clusters (agrégats de Pom1) et la phosphorylation de Pom1. On a la levure wild type et troi types de mutants: 2A, avec deux sites de phosphorylation mutés, 4A, qui en a quatre, et 6A, qui en a six. Comme première chose, on a voulu quantifier les pics des profiles des mutants, pour comprendre si la phosphorylation a un rôle dans le clustering. On a donc travaillé avec la transformée de Fourier, que c’est une somme infinie des fonctions sinus et cosinus ayant comme argument la fréquence. Cette analyse dans R correspond à la fonction spectrum. Au final on a comparés les vecteur avec les différentes hautes fréquences à l’aide d’un test de Student. Les résultats qu’on a obtenus sont des p valeurs significatives, avec un seuil de 0.05, sauf pour le mutant 2A, qui ce n’est pas significativement différent du WT. On a donc conclu que les mutants 4A et 6A sont significativement différents du WT et font moins de clusters. La phosphorylation joue donc un rôle dans le clustering, car moins il peut se phosphoryler, moins il fait des clusters.
Dans un deuxième temps, on a cherché à comprendre quelle implication a le clustering dans la formation du gradient. Pour faire ça on a calculé la quantité de Pom1 dans la cellule. Dans R on s’est servi de la fonction cortical.pom1, qui nous a donné notre superviseur, qui tient en compte la courbe de la cellule. Enfin, on a appliqué aussi des t-tests. On a obtenu des p valeurs pas significatives, donc il n'y a pas plus de Pom1 à la membrane dans les mutants par rapport au WT.
On a expliqué ça par le fait que peut-être que les sites de phosphorylation sont utiles pour s’attacher et se détacher de la membrane.
5 - Conclusions
Notre première conclusion a été que le mode de phosphorylation de Pom1 est trans. Deuxièmement, on a trouvé une corrélation entre la quantité de Tea4 et Pom1, mais pas entre Pom1 et Cdr2. Enfin, la formation de clusters dépend de la phosphorylation. Comme certains résultats n’ont pas été significatifs, d’autres expériences sont nécessaires pour répondre à la question. Pendant ce projet on a pu voir comme des modèles mathématiques peuvent permettre de répondre à des questions biologiques.
Nous remercions chaleureusement notre superviseur Micha Hersch pour son aide tout au long de ce projet.