Difference between revisions of "ProjectYeast"
Sbprm2015 1 (talk | contribs) |
Sbprm2015 1 (talk | contribs) |
||
| Line 86: | Line 86: | ||
'''4 - Clustering de Pom1''' | '''4 - Clustering de Pom1''' | ||
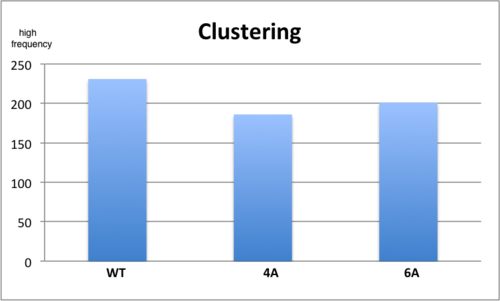
| − | Dans cette partie on va étudier la relation entre la formation de clusters (aggregats de Pom1) et la phosphorylation de Pom1. On a la levure wild type et troi types de mutants: 2A, avec deux sites de phosphorylation mutés, 4A, qui en a quatre, et 6A, qui en a six. Comme première chose, on a voulu quantifier les pics des profiles des mutants, popur comprendre si la phosphorylation a un rôle dans le clustering. On a donc travaillé avec la transfromée de Fourier, que c’est une somme infinie des fonctions sinus et cosinus ayant comme argument la fréquence. Cette analyse dans R corréspond à la fonction ''spectrum''. Au final on a comparés les vecteur avec les différentes hautes fréquences à l’aide d’un test de Student. Les résultats qu’on a obtenus sont des p valeurs significatives, avec un seuil de 0.05, sauf pour le mutant 2A, qui ce n’est pas significativement différent du WT. On a donc conclu que les mutants 4A et 6A sont significativement différents du WT et | + | Dans cette partie on va étudier la relation entre la formation de clusters (aggregats de Pom1) et la phosphorylation de Pom1. On a la levure wild type et troi types de mutants: 2A, avec deux sites de phosphorylation mutés, 4A, qui en a quatre, et 6A, qui en a six. Comme première chose, on a voulu quantifier les pics des profiles des mutants, popur comprendre si la phosphorylation a un rôle dans le clustering. On a donc travaillé avec la transfromée de Fourier, que c’est une somme infinie des fonctions sinus et cosinus ayant comme argument la fréquence. Cette analyse dans R corréspond à la fonction ''spectrum''. Au final on a comparés les vecteur avec les différentes hautes fréquences à l’aide d’un test de Student. Les résultats qu’on a obtenus sont des p valeurs significatives, avec un seuil de 0.05, sauf pour le mutant 2A, qui ce n’est pas significativement différent du WT. On a donc conclu que les mutants 4A et 6A sont significativement différents du WT et font moins de clusters. La phosphorylation joue donc un rôle dans le clustering, car moins il peut se phosphoryler, moins il fait des clusters. |
| − | [[File:clusterin.png|500px|thumb|left| | + | [[File:clusterin.png|500px|thumb|left|formation de clusters par phénotype]] |
| + | |||
| + | |||
| + | |||
| + | |||
| + | |||
| + | |||
| + | |||
| + | |||
| + | |||
| + | |||
| + | |||
| + | |||
| + | |||
| + | |||
| + | |||
| + | Lala | ||
Revision as of 16:48, 23 May 2015
Modeling Gradient Formation in the Fission Yeast
Escher Cécile, Steffe Perrine, Spataro Sofia, Murseli Besa
Superviseur: Hersch Micha
1 - Introduction
Pom1 est une protéine de la levure S. pombe qui permet la division cellulaire. Pom1 régule négativement un activateur de l'entrée en mitose: la protéine kinase Cdr2, qui se situe au niveau de l'équateur de la cellule. Le transport de Pom1 aux pôles de la cellule crée la formation d'un graident de Pom1. Pom1 est recrutée au pôles de la cellule grâce aux microtubules, qui transportent la protéine Tea4 aux extrémités. Cette protéine médie la déphosphorylation de Pom1 en recrutant Disc2. Pom1 se lie à la membrane avec les interaction électrostatiques entre les lipides de la membrane et la séquence de Pom1. Pom1 va diffuser le long de la membrane pour le fait qu'elle est une kinase active et possède plusieurs sites pour la phosphorylation. Son activité d'autophosphorylation diminue son affinité à la membrane, promeut son détachement et ainsi sa diffusion latérale le long de la membrane plasmique. Dans ce projet on va étudier le phenomène de la diffusion de Pom1, en cherchant de l'éxpliquer avec des modèles mathématiques; ensuite on ira aussi étudier les interactions entre Cdr2 et Tea4 et le clustering de Pom1.
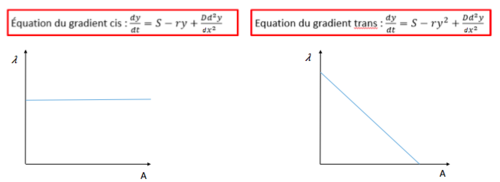
2 - Phosphorylation de Pom1: modèle cis ou trans?
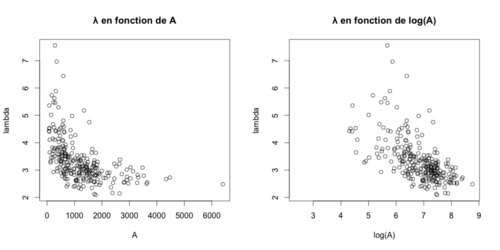
On a analysé avec R les profiles de l'intensité de Pom1 le long de la membrane pour chercher de comprendre si λ (distance à laquelle la concentration de Pom1 diminue d'un facteur donné) dépend de la valeur de A (quantité initiale de Pom1 au temps 0). Si λ ne dépend pas de A le modéle de phosphorylation est dit en cis, au contraire, si λ dépend de A, le modèle est dit en trans et on est en présence d'autophosphorylation. Pour obtenir ces modèles on a travaillé avec des équations différentielles:
Résultats:
Nous avons donc conclu que le mode de phosphrylation de Pom1 est en trans.
3 - Localisation de Cdr2 en fonction de Tea4
Dans cette partie on va étudier la localisation de Cdr2 en fonction de la variabilité de Tea4 auw extrémités de la cellule.
4 - Clustering de Pom1
Dans cette partie on va étudier la relation entre la formation de clusters (aggregats de Pom1) et la phosphorylation de Pom1. On a la levure wild type et troi types de mutants: 2A, avec deux sites de phosphorylation mutés, 4A, qui en a quatre, et 6A, qui en a six. Comme première chose, on a voulu quantifier les pics des profiles des mutants, popur comprendre si la phosphorylation a un rôle dans le clustering. On a donc travaillé avec la transfromée de Fourier, que c’est une somme infinie des fonctions sinus et cosinus ayant comme argument la fréquence. Cette analyse dans R corréspond à la fonction spectrum. Au final on a comparés les vecteur avec les différentes hautes fréquences à l’aide d’un test de Student. Les résultats qu’on a obtenus sont des p valeurs significatives, avec un seuil de 0.05, sauf pour le mutant 2A, qui ce n’est pas significativement différent du WT. On a donc conclu que les mutants 4A et 6A sont significativement différents du WT et font moins de clusters. La phosphorylation joue donc un rôle dans le clustering, car moins il peut se phosphoryler, moins il fait des clusters.
Lala