Difference between revisions of "Genetics of urinary calcium levels"
Sbprm2014 3 (talk | contribs) |
Sbprm2014 3 (talk | contribs) |
||
| (11 intermediate revisions by the same user not shown) | |||
| Line 9: | Line 9: | ||
'''Supervisor''': [[User:Tanguy|Tanguy Corre]] | '''Supervisor''': [[User:Tanguy|Tanguy Corre]] | ||
| − | '''Students''': | + | '''Students''': Athimed El Taher, Elodie Ramella, Valentina Scandella, Emanuele Bulla |
| Line 17: | Line 17: | ||
'''But''' : Identifier les locis du génome qui induisent une variation significative de la concentration en calcium dans l’urine chez les différents individus de l’étude CoLaus | '''But''' : Identifier les locis du génome qui induisent une variation significative de la concentration en calcium dans l’urine chez les différents individus de l’étude CoLaus | ||
| − | '''Aspects biologiques ou médicaux ''': La concentration calcique urinaire varie entre individus. Une partie de cette variation peut être déterminé génétiquement. Dans le cadre de cette étude, nous nous somme justement intéressé à la part de variance expliqué notamment par les SNP | + | '''Aspects biologiques ou médicaux ''': La concentration calcique urinaire varie entre individus. Une partie de cette variation peut être déterminé génétiquement. Dans le cadre de cette étude, nous nous somme justement intéressé à la part de variance expliqué notamment par les SNP. Cette étude peut porter à des aspects intéressants, tels que la détermination génétique des risques d'hyper- ou d'hypocalciurie. Sachant qu'une hypocalciurie chez la femme enceinte peut entraîner de grosses complications à l'accouchement, un génotypage des SNPs responsables de variations de calcium urinaire peut préparer les médecins au pire. On se dirige donc vers une médecine personnalisée. |
'''Outils mathématiques''' : | '''Outils mathématiques''' : | ||
| Line 24: | Line 24: | ||
'''Matériel et méthodes''' : | '''Matériel et méthodes''' : | ||
| + | Pour nos analyses, nous avons employé la base donnée CoLaus contenant entre autre les génotypes de 2,5 millions de SNPs pour plus de 5'000 personne, ainsi que des données phénotypiques (âge, sexe, concentrations urinaires de calcium, créatinine, magnésium, potassium et sodium). | ||
| + | En premier lieu, nous avons fait un contrôle qualité de nos génotypes et de nos phénotypes. Nous avons entre autre remarqué qu’il n’y avait pas d’influence significative de l’âge sur la concentration de calcium urinaire chez les hommes. Par contre, chez les femmes de plus ou moins de 50 ans la différence était notable, très certainement à cause de la ménopause. Ensuite pour que nos données soient utilisables, nous avons vérifié que l’équilibre d’Hardy-Weinberg soit respecté, que le callrate soit supérieur à 0.9, que la fréquence allélique soit supérieure à 0.01 et que le R2 soit supérieur à 0.03 pour les données imputées. | ||
| + | |||
| + | Puis nous avons fait une première analyse d’association sur neuf SNPs candidats trouvés dans des articles traitant de la concentration de calcium urinaire (voir sérique). Nous avons donc appliqué des régression linéaire sur SNPs pour différents ratio : calcium/créatinine, calcium/magnésium, calcium/potassium, calcium/sodium. Après avoir appliqué la correction de Bonferroni, une seule SNP, rs1570669, avait une p-value assez basse (0.0004) pour être en dessous du nouveau seuil de significativité de 0.00139 et cela lorsqu’elle était associée au ration calcium/magnésium. Elle se trouve dans un intron codant pour le cytochrome P450 qui aide la sécrétion des substances dans les urines. | ||
| + | |||
| + | Nous avons ensuite continué en faisant une analyse d’association sur tout le génome. Afin d’analyser les résultats obtenus nous avons représenté visuellement les p-values sous forme de QQplot et de Manhattan plot pour chaque ratio. Ces graphique nous ont permis d’estimer quel seuil de significativité paraissait le plus adapté en fonction des résultats obtenus (en suivant la correction de Bonferroni nous aurions du prendre un seuil α de 10-8, mais nous aurions manqué ainsi plusieurs faux-négatifs) et de voir sur quels chromosomes se trouvait les loci d’intérêt. Nous avons finalement fait un zoom sur les SNPs centrales des ces loci afin de les analyser et de voir sur quel gène ils se trouvaient. | ||
| + | |||
| + | [[File:Capture_d’écran_2014-05-30_à_20.09.53.png|500px]] | ||
| + | Figure 1 : Exemple de Manhattan plot, Ratio Calcium/Créatinine | ||
| + | [[File:Capture_d’écran_2014-05-30_à_20.15.08.png|500px]] | ||
| + | Figure 2 : Exemple de QQplot, Ratio Calcium/Créatinine | ||
'''Résultats principaux''' : | '''Résultats principaux''' : | ||
| Line 30: | Line 41: | ||
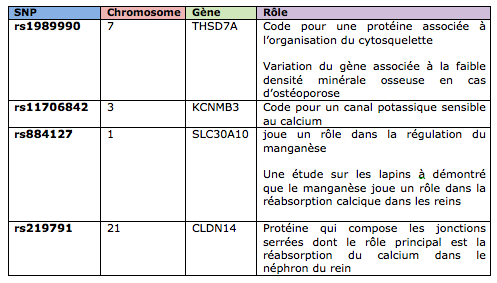
Lors de l'analyse d'association sur tout le génome, nous avons trouvé des SNP sur 4 gènes intéressants. Ces derniers sont regroupé dans un tableau qui se trouve ci-dessous. Le seuil de significativité varie entre 10-5 et 10-8 selon les cas. Nous Avons ajusté le seuil afin d'avoir un minimum de faux négatif. | Lors de l'analyse d'association sur tout le génome, nous avons trouvé des SNP sur 4 gènes intéressants. Ces derniers sont regroupé dans un tableau qui se trouve ci-dessous. Le seuil de significativité varie entre 10-5 et 10-8 selon les cas. Nous Avons ajusté le seuil afin d'avoir un minimum de faux négatif. | ||
| − | [[File:Capture_d’écran_2014-05-29_à_21.58.17.png|thumb|500px]] | + | [[File:Capture_d’écran_2014-05-29_à_21.58.17.png|thumb|500px]] |
| + | |||
| + | '''Conclusions ''': Quatre SNPs on été identifiée pouvant jouer un rôle dans la régulation du calcium urinaire (voir tableau des résultats principaux). Bien sûr ces résultats sont à vérifier dans le contexte où toutes n'ont pas une p-value suffisamment basse. Il se peut donc qu'il y ait des faux négatifs ou des faux positif, ainsi que la suite logique de l'étude serait de faire un replicas sur un nouvelle base de données ou faire une étude fonctionnelle en laboratoire. Cela confirmerait ou dénigrerait les résultats obtenus. | ||
| − | ''' | + | '''Présentation ''':[[Media:Présentation_CalciumUrinaire.pdf]] |
Latest revision as of 19:59, 30 May 2014
Background: Our research group contributed to show that a particular gene is involved in the regulation of calcium serum.
Goal: The goal of this project is to use the same methid, namely Genome-Wide Association Studies to find genes involved in the regulation of urinary calcium.
Mathematical tools: The student will learn the statistics associated with Genome-Wide Association Studies
Biological or Medical aspects: The students will learn how Genome Wide Association Studies can lead to medical discoveries, in particular in the calcium biology
Supervisor: Tanguy Corre
Students: Athimed El Taher, Elodie Ramella, Valentina Scandella, Emanuele Bulla
Contexte : La génétique prend de plus en plus de place dans la recherche avec le séquençage du génome. De plus les études à large échelle fleurissent, comme CoLaus ce qui permet de faire des études d’association génomique (GWAS).
But : Identifier les locis du génome qui induisent une variation significative de la concentration en calcium dans l’urine chez les différents individus de l’étude CoLaus
Aspects biologiques ou médicaux : La concentration calcique urinaire varie entre individus. Une partie de cette variation peut être déterminé génétiquement. Dans le cadre de cette étude, nous nous somme justement intéressé à la part de variance expliqué notamment par les SNP. Cette étude peut porter à des aspects intéressants, tels que la détermination génétique des risques d'hyper- ou d'hypocalciurie. Sachant qu'une hypocalciurie chez la femme enceinte peut entraîner de grosses complications à l'accouchement, un génotypage des SNPs responsables de variations de calcium urinaire peut préparer les médecins au pire. On se dirige donc vers une médecine personnalisée.
Outils mathématiques : Nous avons utilisé le programme Matlab principalement et nous nous somme également aidé du programme R
Matériel et méthodes :
Pour nos analyses, nous avons employé la base donnée CoLaus contenant entre autre les génotypes de 2,5 millions de SNPs pour plus de 5'000 personne, ainsi que des données phénotypiques (âge, sexe, concentrations urinaires de calcium, créatinine, magnésium, potassium et sodium).
En premier lieu, nous avons fait un contrôle qualité de nos génotypes et de nos phénotypes. Nous avons entre autre remarqué qu’il n’y avait pas d’influence significative de l’âge sur la concentration de calcium urinaire chez les hommes. Par contre, chez les femmes de plus ou moins de 50 ans la différence était notable, très certainement à cause de la ménopause. Ensuite pour que nos données soient utilisables, nous avons vérifié que l’équilibre d’Hardy-Weinberg soit respecté, que le callrate soit supérieur à 0.9, que la fréquence allélique soit supérieure à 0.01 et que le R2 soit supérieur à 0.03 pour les données imputées.
Puis nous avons fait une première analyse d’association sur neuf SNPs candidats trouvés dans des articles traitant de la concentration de calcium urinaire (voir sérique). Nous avons donc appliqué des régression linéaire sur SNPs pour différents ratio : calcium/créatinine, calcium/magnésium, calcium/potassium, calcium/sodium. Après avoir appliqué la correction de Bonferroni, une seule SNP, rs1570669, avait une p-value assez basse (0.0004) pour être en dessous du nouveau seuil de significativité de 0.00139 et cela lorsqu’elle était associée au ration calcium/magnésium. Elle se trouve dans un intron codant pour le cytochrome P450 qui aide la sécrétion des substances dans les urines.
Nous avons ensuite continué en faisant une analyse d’association sur tout le génome. Afin d’analyser les résultats obtenus nous avons représenté visuellement les p-values sous forme de QQplot et de Manhattan plot pour chaque ratio. Ces graphique nous ont permis d’estimer quel seuil de significativité paraissait le plus adapté en fonction des résultats obtenus (en suivant la correction de Bonferroni nous aurions du prendre un seuil α de 10-8, mais nous aurions manqué ainsi plusieurs faux-négatifs) et de voir sur quels chromosomes se trouvait les loci d’intérêt. Nous avons finalement fait un zoom sur les SNPs centrales des ces loci afin de les analyser et de voir sur quel gène ils se trouvaient.
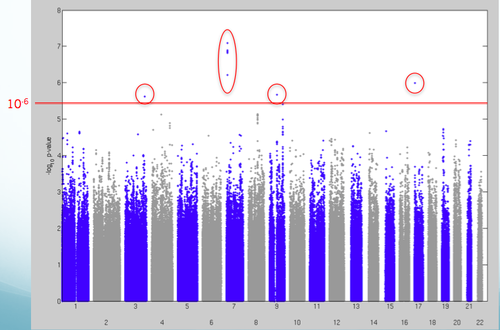 Figure 1 : Exemple de Manhattan plot, Ratio Calcium/Créatinine
Figure 1 : Exemple de Manhattan plot, Ratio Calcium/Créatinine
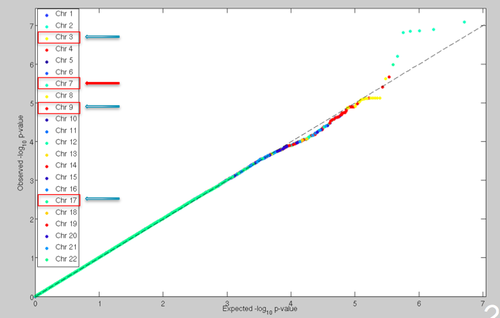 Figure 2 : Exemple de QQplot, Ratio Calcium/Créatinine
Figure 2 : Exemple de QQplot, Ratio Calcium/Créatinine
Résultats principaux :
Lors de l'analyse d'association sur tout le génome, nous avons trouvé des SNP sur 4 gènes intéressants. Ces derniers sont regroupé dans un tableau qui se trouve ci-dessous. Le seuil de significativité varie entre 10-5 et 10-8 selon les cas. Nous Avons ajusté le seuil afin d'avoir un minimum de faux négatif.
Conclusions : Quatre SNPs on été identifiée pouvant jouer un rôle dans la régulation du calcium urinaire (voir tableau des résultats principaux). Bien sûr ces résultats sont à vérifier dans le contexte où toutes n'ont pas une p-value suffisamment basse. Il se peut donc qu'il y ait des faux négatifs ou des faux positif, ainsi que la suite logique de l'étude serait de faire un replicas sur un nouvelle base de données ou faire une étude fonctionnelle en laboratoire. Cela confirmerait ou dénigrerait les résultats obtenus.
Présentation :Media:Présentation_CalciumUrinaire.pdf