Difference between revisions of "Genetics of eating patterns"
| (11 intermediate revisions by the same user not shown) | |||
| Line 24: | Line 24: | ||
Suite à la décomplexification des phénotypes par l’analyse en composante principale, des régressions linéaires ont été appliquées sur les 2.5 millions de SNPs. Les P-values obtenues grâce aux régressions linéaires ont permis de représenter et visualiser les résultats d’associations (p-values) sous forme de Manhattan plots et de QQ plots. A noter que ces résultats d’associations ont été corrigés par le nombre de tests indépendants réalisés. | Suite à la décomplexification des phénotypes par l’analyse en composante principale, des régressions linéaires ont été appliquées sur les 2.5 millions de SNPs. Les P-values obtenues grâce aux régressions linéaires ont permis de représenter et visualiser les résultats d’associations (p-values) sous forme de Manhattan plots et de QQ plots. A noter que ces résultats d’associations ont été corrigés par le nombre de tests indépendants réalisés. | ||
Une SNP significative a été étudiée de manière plus approfondie par le biais d’un locus zoom et de la banque de données NCBI qui ont permis de déterminer si cette SNP se trouvait dans un gène, la fonction de ce gène et les études qui étaient associées. | Une SNP significative a été étudiée de manière plus approfondie par le biais d’un locus zoom et de la banque de données NCBI qui ont permis de déterminer si cette SNP se trouvait dans un gène, la fonction de ce gène et les études qui étaient associées. | ||
| + | |||
| + | [[File:Manhattan plot.jpg]] | ||
| + | Figure 1 : Manhattan plot de la composante principale 2 | ||
| + | |||
| + | [[File:Locus Zoom.jpg]] | ||
| + | Figure 2 : Locus Zoom de la SNP significative | ||
| + | |||
Une fois la SNP significative déterminée, une analyse plus détaillée des phénotypes a été réalisée, en considérant cette fois les 97 aliments, permettant d’établir l’influence de cette SNP sur la consommation des aliments. | Une fois la SNP significative déterminée, une analyse plus détaillée des phénotypes a été réalisée, en considérant cette fois les 97 aliments, permettant d’établir l’influence de cette SNP sur la consommation des aliments. | ||
| Line 43: | Line 50: | ||
Etudiants : Lucie Bidlingmeyer, Phanie Bidlingmeyer, Yu Larpin | Etudiants : Lucie Bidlingmeyer, Phanie Bidlingmeyer, Yu Larpin | ||
| − | Présentations : | + | Présentations :[[Media:Présentation Finale.pptx]] |
Références : http://www.ncbi.nlm.nih.gov/projects/SNP/snp_ref.cgi?rs=7623540, http://www.genome.gov/gwastudies/index.cfm?pageid=26525384#searchForm | Références : http://www.ncbi.nlm.nih.gov/projects/SNP/snp_ref.cgi?rs=7623540, http://www.genome.gov/gwastudies/index.cfm?pageid=26525384#searchForm | ||
Latest revision as of 13:23, 5 June 2013
Etude de la génétique des préférences alimentaires
Contexte : Actuellement les liens entre la génétique et les habitudes alimentaires sont peu connus car peu d’études génétiques ont été réalisées sur ce sujet.
Le recensement de CoLaus a permis de récolter des données à large échelle concernant les individus de la région lausannoise, comme par exemple les habitudes alimentaires. Ainsi ces données ont de grandes chances d’apporter des résultats inédits.
But : Le but de ce projet était de faire une étude d’associations sur l’ensemble du génome (GWAS) pour les habitudes alimentaires, afin de tenter de démontrer qu’il existe une composante génétique liée à la variabilité des préférences alimentaires.
Aspects biologiques ou médicaux :
L’intérêt de cette étude est de mieux comprendre la base génétique des habitudes alimentaires qui peuvent entraîner des dérèglements du comportement (obésité, anorexie,…). Une meilleure connaissance de ce sujet pourrait permettre une médication personnalisée.
Outils mathématiques :
Utilisation de Matlab, R
Matériel et méthodes :
Les données employées dans cette recherche, provenant de CoLaus, sont les génotypes (2,5 millions de SNPs mesurées et imputées) et les phénotypes (les fréquences de consommation de 97 aliments).
Une analyse en composante principale a été effectuée à partir des 97 phénotypes. Les 10 premières principales composantes ont été retenues, expliquant 37.5% de la variabilité phénotypique.
Suite à la décomplexification des phénotypes par l’analyse en composante principale, des régressions linéaires ont été appliquées sur les 2.5 millions de SNPs. Les P-values obtenues grâce aux régressions linéaires ont permis de représenter et visualiser les résultats d’associations (p-values) sous forme de Manhattan plots et de QQ plots. A noter que ces résultats d’associations ont été corrigés par le nombre de tests indépendants réalisés. Une SNP significative a été étudiée de manière plus approfondie par le biais d’un locus zoom et de la banque de données NCBI qui ont permis de déterminer si cette SNP se trouvait dans un gène, la fonction de ce gène et les études qui étaient associées.
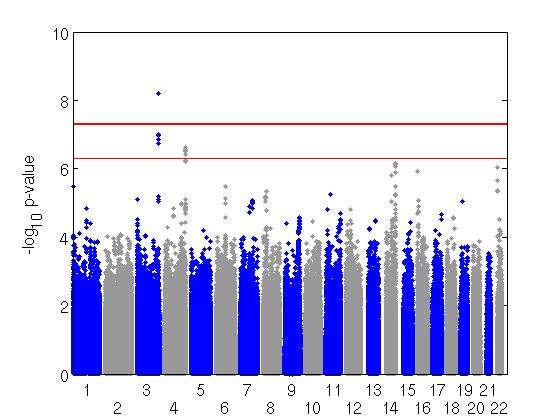 Figure 1 : Manhattan plot de la composante principale 2
Figure 1 : Manhattan plot de la composante principale 2
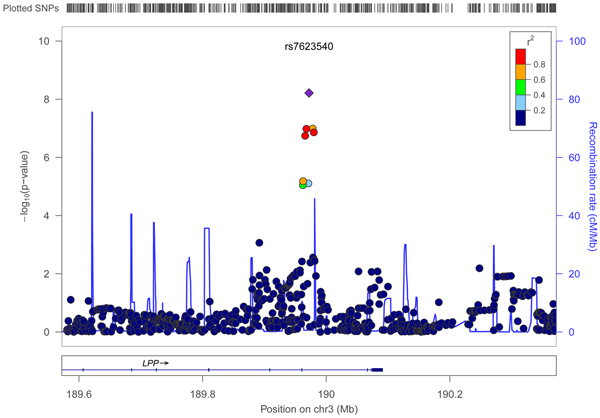 Figure 2 : Locus Zoom de la SNP significative
Figure 2 : Locus Zoom de la SNP significative
Une fois la SNP significative déterminée, une analyse plus détaillée des phénotypes a été réalisée, en considérant cette fois les 97 aliments, permettant d’établir l’influence de cette SNP sur la consommation des aliments.
Résultats principaux :
Une SNP significative au niveau du génome a été trouvée : rs7623540. Elle se situe sur le chromosome 3, dans le gène LPP (lipoma-preferred partner) qui a un rôle dans l’adhésion et la mobilité cellulaire. Des études avaient déjà associé l’obésité, la maladie cœliaque et d’autres maladies auto-immunes à ce gène.
Selon nos résultats, la présence de cette SNP est associée à une faible consommation de beefsteak de cheval et de croissants.
D’autres SNPS ne dépassant pas le seuil de significativité au niveau du génome ont été analysées sur l’ensemble des composantes principales sans donner de résultats concluants.
Conclusions :
La présence d’une SNP qui influence les préférences alimentaires a été découverte (rs7623540) dans le gène LPP.
L’étape suivante de ce projet pourrait être de répliquer les résultats obtenus dans une autre cohorte, et de planifier des expérimentations (par exemple animales) pour obtenir une validation fonctionnelle.
Assistants : Diana Marek, Tanguy Corre
Etudiants : Lucie Bidlingmeyer, Phanie Bidlingmeyer, Yu Larpin
Présentations :Media:Présentation Finale.pptx
Références : http://www.ncbi.nlm.nih.gov/projects/SNP/snp_ref.cgi?rs=7623540, http://www.genome.gov/gwastudies/index.cfm?pageid=26525384#searchForm